La biométhanisation publié le 23/05/2022
2.3 Écosystème microbien du digestat
Le digestat présent dans le méthaniseur est un écosystème complexe très hétérogène par sa composition, et qui rassemble un grand nombre de microorganismes variés.
Les nombreux microorganismes impliqués dans cette digestion anaérobie, sont en relation étroite entre eux car les réactions des uns fournissent les substrats des autres.
La composition de la population microbienne présente dans les méthaniseurs n’est généralement pas entièrement identifiée et elle varie d’un digestat à l’autre. Cependant les microorganismes présents sont des microorganismes, en principe, thermophiles ou mésophiles et ils appartiennent aux domaines que sont les Bacteria et les Archaea .
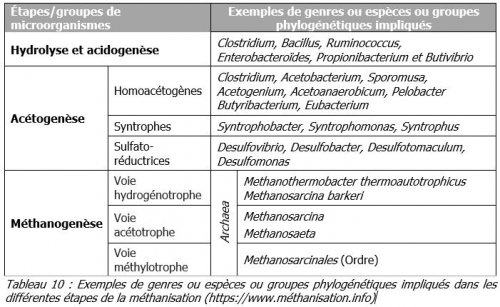
Les microorganismes issus de ces deux domaines impliqués dans la méthanisation, peuvent être classés en fonction des étapes dans lesquelles ils interviennent généralement.
On va décrire ainsi 3 groupes :
- Les microorganismes hydrolytiques et fermentatifs qui réalisent les étapes d’hydrolyse et d’acidogénèse
- Les microorganismes acétogènes impliqués dans l’acétogenèse.
- Les microorganismes méthanogènes sont ceux qui interviennent dans la dernière phase du processus aboutissant à la synthèse de méthane. Il est à signaler que ces méthanogènes sont tous des Archaea . Une cinquantaine d’espèces de ces méthanogènes ont déjà été décrites. Elles sont toutes anaérobies. Cependant elles appartiennent à des groupes phylogéniques pouvant être différents (différents clades).
Ces trois communautés doivent constituer un écosystème symbiotique. Un équilibre doit être trouvé pour que chaque microorganisme puisse se développer et synthétiser ces produits. De plus, les produits des uns ne doivent pas inhiber le développement des autres.
Les conditions optimales de croissance des bactéries hydrolytiques et acidogènes sont relativement les mêmes avec une gamme de pH commune aux alentours de 4,5 à 6,5. Alors que les 2 autres étapes nécessitent des conditions optimales avec un pH plus élevé (6,8 à 7,5). L’ensemble de ces microorganismes évoluant dans le même écosystème, les conditions ne sont a priori jamais optimales pour l’ensemble des familles. Le pH se situe naturellement autour de 7-7,5 et sauf cas particulier, aucune régulation de pH n’est à effectuer, excepté en cas de réelle acidose (la chute du pH est due à une accumulation d’acides provoquant une interruption de la production de biogaz).
La liste exacte des différentes espèces de microorganismes opérant dans chacune de ces étapes est, pour la plupart des cas, mal définie car leur culture est difficile par des techniques classiques de laboratoire, notamment par le fait que ces souches soient anaérobies (certaines bactéries de l’étape d’hydrolyse peuvent tolérer la présence d’oxygène) et que leur nombre et leurs variétés soient importants. De plus, les interactions entre les espèces peuvent aussi en modifier leurs propriétés ce qui pourrait mal orienter l’identification par comparaison avec une souche pure.
Il est à noter que les microorganismes présents dans les digesteurs dépendent du type de substrat présent dans le digesteur. L’alimentation par du lisier conduit à un haut taux d’ammoniac et d’acides gras volatils faisant apparaitre de façon majoritaire des Methanosarcina, alors qu’une alimentation par des boues, baisse les taux d’ammoniac et d’acides gras volatils et favorise la présence de Methanosaeta (Karakashev et al. 2005).
La connaissance des différentes espèces est donc un travail très complexe du fait du nombre d’espèces présentes. Des travaux de métagénomique et métabolomique, de plus en plus nombreux, ont pour objectif de les identifier et de comprendre leur rôle. En effet, la connaissance des espèces permettrait une compréhension des mécanismes impliqués et ainsi un meilleur contrôle du processus dans sa globalité (St-Pierre and Wright, 2014 ; Lu et al., 2013 Schluter et al., 2008)
2.4 Différentes pistes d’optimisation
a. Études méta-omiques
Pour améliorer et optimiser la production et les process de méthanisation, il est probablement intéressant de connaitre la composition exacte en microorganismes responsables des diverses réactions biochimiques mais aussi de connaître les interactions entre les espèces et la fonction de chacune dans cette communauté. De nouvelles approches de biologie moléculaire appuyées par la bioinformatique permettent d’améliorer nos connaissances sur ces populations et leurs fonctions au sein de ces écosystèmes. La métagénomique, la métatranscriptomique, la métaprotéomique ou encore la métabolomique permettent, ainsi, de révéler la microbiologie de ces systèmes complexes en lien avec leurs potentialités fonctionnelles.
Du séquençage haut-débit (NGS pour Next-Generation Sequencing) et de la qPCR sur ces populations et leurs milieux permettent, maintenant, d’accéder finement aux capacités fonctionnelles des écosystèmes de méthanisation.
A propos des études métagénomiques, deux stratégies sont possibles :
- Le séquençage global qui consiste à séquencer et analyser l’ensemble des séquences présentes dans l’échantillon de digestat.
- Le séquençage ciblé 16S qui est un séquençage ciblé du gène codant l’ARNr 16S mettant en évidence la représentation et l’abondance des différentes espèces de Bacteria ou Archaea d’un digestat donné.
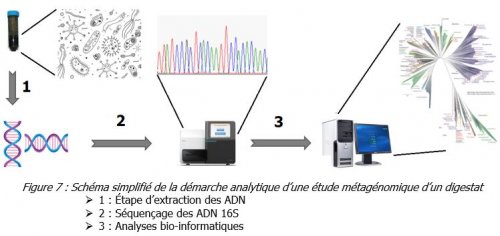
Cette deuxième technique est la plus utilisée car moins coûteuse, elle est souvent suffisante pour identifier les microorganismes présents.
Cette technique comprend différentes étapes (figure 7) comme l’extraction de l’ensemble des ADN génomiques présents dans l’échantillon, la détection et l’analyse des gènes codant l’ARN ribosomique 16S, présent chez ces Procaryotes. L’étude des variations génétiques sur ce gène permettent une analyse phylogénétique. Dans la pratique, on n’analyse pas la totalité de la séquence 16S (1500 pb) mais qu’une partie, en général la région V3-V4. Cette région est souvent suffisante pour identifier le microorganisme jusqu’au genre.
b. Pré-traitements
Bien souvent, les biodégradations par les différents microorganismes présents dans les digesteurs ne sont pas totales. Par exemple, la partie lignocellulosique des déchets organiques végétaux est difficilement biodégradable. La lignine est une molécule organique extrêmement résistante à la dégradation biologique mais certains champignons sont capables de la dégrader principalement par des réactions d’oxydation.
Au-delà des méthodes classiques de broyage, des traitements thermochimiques qui permettent de rendre plus rapidement les substrats (la cellulose) accessibles, se développent de nouveaux pré-traitements qui permettent d’optimiser ces productions. On peut évoquer, notamment, la réalisation de traitements enzymatiques (à partir de ligninases extraites des champignons) qui permettent de pré-dégrader la lignine en la décrochant de la cellulose. On peut citer la Lignine peroxydase (LiP) ou bien la Manganèse peroxydase (MnP) extraite d’un champignon (Phanerochaete chrysosporium).
c. Étude du virome
Parmi les voies d’amélioration possibles, l’étude de virus des microorganismes méthanogènes semblent être une voie prometteuse et particulièrement celle des virus des archées méthanogènes. En effet, ces derniers sont mal connus et mal définis encore aujourd’hui et leur utilisation éventuelle comme vecteurs de transduction pourrait permettre l’optimisation du rendement de la production du biogaz ou bien la réalisation de biocontrôles à des fins de bonne conduite du process, comme c’est déjà le cas dans d’autres domaines (médical, agro-alimentaire, épuration des eaux usées). Les virus de bactéries et d’archées pourraient être utilisés pour favoriser les voies de fermentation les plus optimales et établir des populations plus standardisées et efficientes. Ces virus pourraient donc servir d’outils ciblant spécifiquement certains groupes fonctionnels microbiens.
De plus, la connaissance de ces virus pourrait aider à les neutraliser en cas d’effets parasitaires négatifs sur les microorganismes d’intérêts présents dans les digestats.
Le projet VIRAME proposé par l’université Paris Saclay, a pour objectif de caractériser, in situ, le contenu génomique de virus d’archées méthanogènes dans les bioprocédés anaérobies de valorisation des déchets organiques. Ce projet souhaite établir le lien entre un virus et son hôte au sein d’écosystèmes complexes comme les digestats. Différentes techniques de biologie moléculaire (méta-omiques, isotopie, en particulier « Stable isotope probing » SIP) et des analyses in silico classiques (génomique comparative) et spécifiques (analyse de CRISPR spacers, de provirus, composition en k-mers), ont pour objectif de définir les liens entre les voies de méthanogenèse réalisées par les archées et la présence des virus infectant ces archées.