Lifemap publié le 10/10/2016 - mis à jour le 21/12/2022
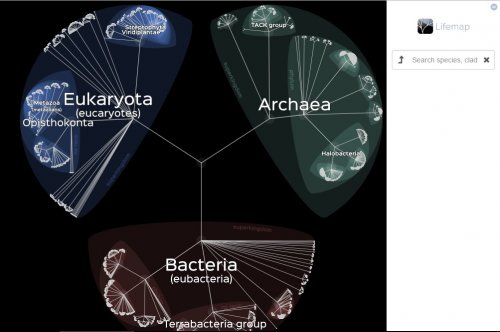
L'arbre de vie intéractif
Le concept utilisé dans LifeMap est similaire à celui utilisé en cartographie avec des outils en ligne comme Google Maps © ou Open Street Maps : l’exploration se fait en zoomant pour obtenir des détails ou en dézoomant pour obtenir une vue panoramique.
Tous les nœuds de l’arbre sont cliquables. Il affiche des informations (description et image) concernant les taxons (extrait de la page Wikipedia, le cas échéant).
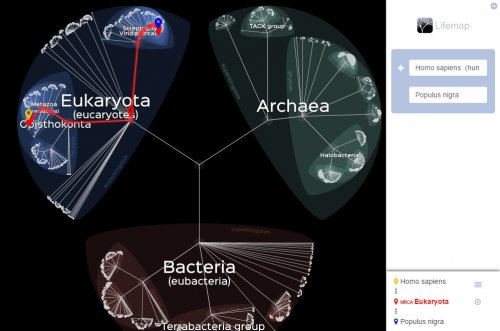
Les itinéraires entre les taxons peuvent être calculés en remplissant le champ de recherche à droite.
Lors du remplissage des deux nouveaux champs de recherche, la route est tracée entre les taxons et l’ancêtre commun le plus récent (MRCA : Most Recent Common Ancestor) des deux groupes ou espèces.
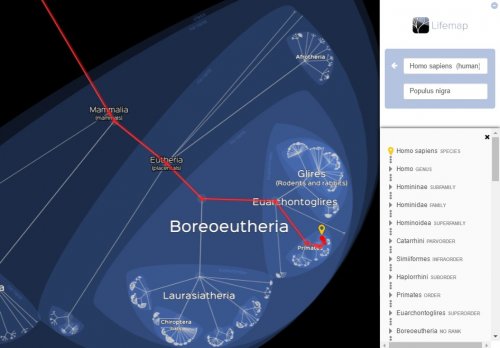
L’utilisateur obtient également l’accès à une liste de tous les noeuds que l’on rencontre dans la voie d’un groupe à l’autre. Cette liste est cliquable, et le zoom ou le dézoom se fait automatiquement.
(1) lifemap a été écrit par damien de Vienne, chercheur au cnrs, travaillant au laboratoire de biométrie et biologie evolutive (lbbe) à lyon avec le soutien du département de recherche en informatique (en particulier stephane delmotte et bruno spataro).
Une version « pro » de lifemap, dénommée lifemap ncbi version, dédiée aux chercheurs en génomique, génétique et biologie évolutionniste existe aussi. Elle montre la taxinomie exhaustive du NCBI (plus de 1,1 million de taxons) et permet (i) un accès facile aux pages web du site, (ii) la visualisation et le nombre de génomes complètement séquencés et (iii) la possibilité de télécharger chaque sous - arbre pour chaque noeud.